1. Motivate
본 내용에서 다루고자 하는 주 내용은 count time series를 일반화 선형 모형(Generalized Linear Model: GLM) 접근방법을 이용해 모델링하여 temporal correlation에 대한 파악과 count data Yt에 대한 이산형 주변 분포(discrete marginal distribution)를 구하고자 합니다. Count time series가 포아송 조건부 분포(Poisson conditional distribution)와 음이항 조건부 분포(Negative Binomial conditional distribution)를 따르는 경우에 대해 다룹니다.
2. Background
본 section 2에서는 포아송 분포(Poisson distribution)와 음이항 분포(Negative Binomial) 및 일반화 선형 모형(Generalized Linear Model: GLM)에 대해 살펴보고자 합니다.
2.1 Poisson Distribution
포아송 분포는 단위 시간 안에 특정 사건이 일어난 횟수에 대해 모델링할 때 사용됩니다.
Y∼Pois(λ),λ>0,y=0,1,2,⋯
단위 시간 안에 특정 사건이 일어날 횟수에 대한 기댓값을 λ라고 했을 때 그 사건이 y회 일어날 확률은 다음과 같습니다.
P(Y=y)=exp(−λ)λyy!,E[Y]=Var[Y]=λ
포아송 분포의 경우 기대값과 분산이 모두 λ로 두 값이 같다는 중요한 특성이 존재합니다.
2.2 Negative Binomial Distribution
만약 Var(Y)>E[Y]인 경우는 과대산포(over-dispersion)에 대해서 고려하여야 합니다. 이에 대하여 감마 분포(Gamma distribution)를 따르는 z가 주어졌을 때 Y의 조건부 분포(conditional distribution)는 포아송 분포인 포아송 감마 혼합 분포(Poisson Gamma Mixture Distribution)을 고려할 수 있습니다.
Y|z∼Pois(λz)z∼Gamma(a,b),
- 형태모수(shape parameter) a>0, 척도모수(scale parameter) b>0
- E(Y|z)=Var(Y|z)=λz, E(z)=ab,Var(z)=ab2
Y의 평균이 λ가 되도록 보장하기 위해서(E[Y]=λ), 감마분포의 파라미터 각각을 a=ν와 b=1ν로 정의하면 Y의 주변 분포는 음이항 분포를 따릅니다.
Y∼NegBin(λ,ν)
f(y;ν)=∫∞0f(y|z)f(z;ν)dz=Γ(ν+y)Γ(y+1)Γ(ν)(νν+λ)ν(λν+λ)y
Y의 평균과 분산은 각각 E[Y]=λ, Var[Y]=λ+λ2/ν이며 분산을 확인해보면 λ에 식 λ2/ν 이 추가된 것을 확인할 수 있습니다. 즉, Var[Y]>E[Y]로 과대산포에 대해서 고려하였음을 알 수 있습니다.
2.3 Generalized Linear Model
일반화 선형 모형(GLM)이란 X=(X1,X2,⋯,Xp)가 주어졌을 때 반응변수 Y의 기댓값 E[Y]의 함수와 독립변수 X와의 선형관계를 모델링한 회귀 모델(regression model)입니다.
일반화 선형 모형의 경우, 반응변수의 분포는 정규분포(Normal distribution)뿐만 아니라 이항 분포(Binomial Distribution), 포아송 분포 등도 사용 가능합니다.
g(E[Y])=Xβ
일반화 선형 모형(GLM)의 구성 요소는 다음 3가지가 존재합니다.
- 확률 요소(Random Component): y=(y1,y2,⋯,yn)T
- 반응 변수 y의 확률분포는 지수족(Exponential Family)에 속하는 분포이어야 합니다.
- 선형 예측자(Link Predictor): Xβ
- 선형 예측자는 design Matrix X와 Parameter β의 선형 결합입니다.
- X∈Rn×p, β=(β1,β2,⋯,βp)T
- 연결 함수(Link Function) : g(⋅)
- g(⋅)는 선형 예측자 Xβ와 반응 변수의 기댓값 E[Y]을 연결해주는 함수입니다.
- 연결 함수의 종류에는 항등함수(Identity function), 로짓/시그모이드 함수(Logit/Sigmoid function) 그리고 로그 함수(log function)가 있습니다.
3. Linear Models For Count Time Series
주 내용인 본 section 3에서는 count time series를 일반화 선형 모형(GLM)으로 적합하는 방법에 대해 설명하고자 합니다.
3.1 Poisson Regression Models For Count Time Series
반응변수 {Yt}는 count time series이며 서로 상관되어있기(correlated) 때문에 이전 관측치 Ft−1={Yt−1,Yt−2,⋯,λ0}가 주어졌을 때의 조건부 분포 Yt|Ft−1을 다음과 같이 포아송 자기회귀이동평균(Autoregressive Moving-average : ARMA(1,1)) 모형으로 적합합니다.
Yt|Ft−1∼Poisson(λt)λt=d+a1λt−1+b1Yt−1,t≥1
- E[Yt|Ft−1]=Var[Yt|Ft−1]=λt
- Generalized Linear Model for Count Time Series
- g(E[Yt|Ft−1])=g(λt)=d+a1λt−1+b1Yt−1
- Link function g(⋅)=Identity Function
조건부 기댓값에 대한 함수는 항등 함수를 사용했으며 조건부 기댓값 λt는 0보다 커야하므로 d,{a1,b1}>0의 제약조건이 필요합니다. 이제 식 (1)을 이용하여 Yt의 주변 분포를 구하고자 합니다.
Yt=λt+(Yt−λt)=λt+ϵt=d+(a1+b1)Yt−1+ϵt−a1ϵt−1=d+(a1+b1)(d+(a1+b1)Yt−2+ϵt−1−a1ϵt−2)+ϵt−a1ϵt−1=⋯=d1−(a1+b1)+∞∑i=0(a1+b1)iϵt−i−a1∞∑j=0(a1+b1)jϵt−j−1
Yt의 모형은 자기회귀이동평균(ARMA(1,1)) 모형임을 알 수 있으며 정상성(stationary)을 보장하기 위해서 0<a1+b1<1의 제약조건이 필요합니다. ϵt의 경우 평균, 공분산이 모두 시간 t에 의존하지 않기 때문에 White Noise(WN) Process를 따르는 것을 알 수 있습니다.
- E[ϵt]=E[yt−λt]=E[E[yt−λt|Ft−1]]=0
- Var[ϵt]=Var[E[ϵt|Ft−1]]+E[Var[ϵt|Ft−1]]=E[λt]=E[Yt]
- Cov(ϵt,ϵt+h)=E[ϵtϵt+h]=E[ϵtE[ϵt+h|Ft+h−1]]=0
식 (2)를 이용하여 Yt의 평균 및 공분산, 상관계수를 구하면 다음과 같습니다.
- E[Yt]=d1−(a1+b1)=μ
- Cov(Yt,Yt+h)={(1−(a1+b1)2+b21)μ1−(a1+b1)2if h=0b1(1−a1(a1+b1))(a1+b1)h−1μ1−(a1+b1)2if h≥1
- b1=0이면 E[Yt]=Var[Yt]인 반면 b1>0에 대해서는 Var[Yt]>E[Yt]입니다.
즉, b1>0인 경우 과대산포가 존재하며 Yt의 주변 분포는 포아송 분포를 따르지 않음을 알 수 있습니다
- b1=0이면 E[Yt]=Var[Yt]인 반면 b1>0에 대해서는 Var[Yt]>E[Yt]입니다.
- Corr(Yt,Yt+h)=b1(1−a1(a1+b1))(a1+b1)h−1(1−(a1+b1)2)
- 제약조건 {a1≥0,b1≥0,0<a1+b1<1}이 존재하기 때문에 항상 상관계수 Corr(Yt,Yt+h)>0입니다. 즉, 오직 Yt끼리 양의 상관관계를 가졌을 때만 연결 함수 g(⋅)를 항등함수로 사용가능합니다. 만약, 음의 상관관계를 가진 Yt를 고려할 경우 연결 함수 g(⋅)는 로그 함수를 사용하면 됩니다.
3.2 Negative Binomial Models For Count Time Series
b1>0인 경우 과대산포가 존재하는 것을 고려하기 위해 포아송 감마 혼합 모델을 사용합니다. 이전 관측치 Ft−1={Yt−1,Yt−2,⋯,λ0}가 주어졌을 때의 조건부 분포 Yt|Ft−1을 다음과 같이 음이항 자기회귀이동평균(Autoregressive Moving-average : ARMA(1,1)) 모형으로 적합합니다.
Yt|Ft−1∼NegBin(λt,ν)λt=d+a1λt−1+b1Yt−1,t≥1
Yt의 조건부 분포의 평균 및 분산은 각각 E[Yt|Ft−1]=λt와 Var[Yt|Ft−1]=λt+λ2t/ν 같으며 λt에 과대산포를 고려한 식 λ2t/ν이 추가된 것을 확인할 수 있습니다.
식 (3)을 이용해 Yt의 주변 분포를 구하면 다음과 같습니다.
Yt=d+(a1+b1)Yt−1+ϵt−a1ϵt−1=d1−(a1+b1)+∞∑i=0(a1+b1)iϵt−i−a1∞∑j=0(a1+b1)jϵt−j−1
ϵt의 평균, 공분산은 다음과 같으며 포아송과는 다르게 공분산에 식 E[λ2t]/ν이 존재합니다.
- E[ϵt]=0,Cov[ϵt,ϵt+h]=0
- Var[ϵt]=Var[E[ϵt|Ft−1]]+E[Var[ϵt|Ft−1]]=E[λt+λ2tν]=E[Yt]+E[λ2t]ν
식 (4)를 이용하여 Yt의 평균 및 공분산, 상관계수를 구하면 다음과 같으며 평균과 상관계수는 Yt|Ft−1가 포아송 분포를 따를 때와 같은 반면 공분산의 경우 더 큰 값을 가지는 것을 확인할 수 있습니다. 다만 ν→∞(1/ν→0)라면, 음이항 분포는 포아송 분포로 수렴합니다.
- E[Yt]=d1−(a1+b1)=μ
- Cov(Yt,Yt+h)={(1−(a1+b1)2+b21)1−(a1+b1)2−b21ν(μ+μ2ν)if h=0b1(1−a1(a1+b1))(a1+b1)h−11−(a1+b1)2−b21ν(μ+μ2ν)if h≥1
- Corr(Yt,Yt+h)=b1(1−a1(a1+b1))(a1+b1)h−1(1−(a1+b1)2)
4. Model Parameter Estimation
Section 4에서는 로그 우도 가능도 함수를 사용해 모수를 추정하는 방법을 소개하고자 합니다. 지금까지는 ARMA(1,1)에 대해 고려하였다면 모수 추정 부분에서는 더 일반화된 모형 ARMA(p,q)를 고려하고자 합니다.
λt=d+q∑i=1aiλt−i+p∑j=1bjYt−j
4.1 Model Parameter Estimation for Linear Poisson Model
먼저 Yt의 조건부 분포가 포아송 분포을 따르는 경우에 대해 로그 가능도 함수는 다음과 같습니다.
Yt|Ft−1∼Poisson(λt),λt=d+q∑i=1aiλt−i+p∑j=1bjYt−j
l(θ)=N∑t=1(−λt(θ)+ytlog(λt(θ))−log(yt!)),θ=(d,a1,⋯,aq,b1,⋯,bp)
θ는 regression parameter이며 조건부 기댓값 λt는 θ의 의존하기 때문에 시간 t에 대한 평균 함수로 정의합니다. 만약, E[Yt]=Var[Yt]인 경우, 즉, Yt가 포아송 분포를 따르는 경우 θ의 최대가능도추정량(Maximum Likelihood Estimator: MLE) ˆθ은 로그 가능도 함수를 최대화하는 θ 값입니다.
ˆθ=argmaxθ∈Θl(θ),Θ={d>0,ai≥0,bi≥0,∑iai+∑jbj<1}
4.2 Model Parameter Estimation for Linear NB Model
Yt의 조건부 분포가 음이항 분포을 따르는 경우에 대해 로그 가능도 함수는 다음과 같습니다.
Yt|Ft−1∼NegBin(λt),λt=d+q∑i=1aiλt−i+p∑j=1bjYt−j
l(θ;ν)=N∑t=1[log(Γ(yt+ν)Γ(yt+1)Γ(ν))+νlog(νν+λt(θ))+ytlog(λt(θ)ν+λt(θ))],θ=(d,a1,⋯,aq,b1,⋯,bp)
추정해야 할 파라미터는 regression parameter θ 뿐만 아니라 dispersion parameter ν도 존재합니다. θ가 ν에 의존하지 않기 때문에 기존의 최대 우도 가능도 방법을 사용하지 않고 Quasi conditional maximum likelihood 방법을 사용하며 크게 2단계로 이루어집니다.
첫번째 단계, regression parameter θ를 추정하기 위해 포아송 분포 기반 quasi maximum likelihood를 사용합니다. 제약 조건 하에서 conditional quasi log likelihood function은 다음과 같으며 l(θ)를 최대화하는 ˆθ는 Quasi maximum likelihood estimator(QMLE) 입니다.
l(θ)=n∑t=1log(pt(yt;θ))=n∑t=1(ytlog(λt(θ))−λt(θ)),ˆθ=argmaxθ∈Θl(θ)
pt(yt;θ)=P(Yt=y|Ft−1)=λytexp(−λt)y!,t=0,1,2,⋯
두번째 단계, dispersion parameter ˆν은 독립적으로 추정됩니다. QMLE ˆθ를 이용하여 조건부 기댓값 ˆλ=λt(ˆθ)을 계산합니다. Christou and Fokianos(2014)에 따르면, 값은 피어슨 카이제곱(Pearson's χ2) 통계량을 기반으로 된 식 (5)를 만족하는 해(solution)를 ˆν 값으로 정의합니다.
N−(p+q+1)=N∑i=1(Yt−^λt)2^λt(1+^λtν)
ˆν=(1NN∑t=1[(Yt−^λt)2−^λt]^λt2)−1
5. Experiment
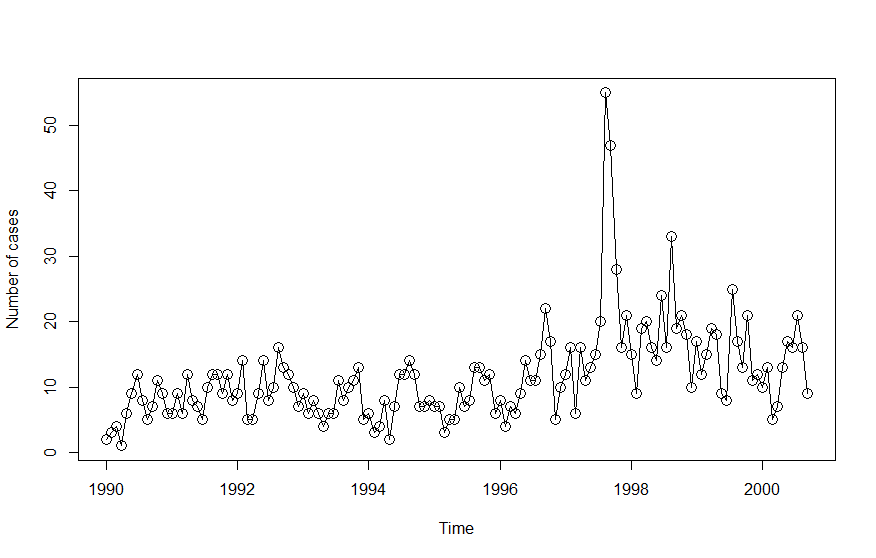
R에서 제공하는 Campy Dataset은 캐나다에서 1990년 1웗루터 2000년 10월까지 한달을 단위로 campylobacterosis 질병에 걸린 횟수에 대한 자료입니다. 일반화 선형 모형(GLM)에 대해서 모형화는 식 (6)과 같습니다.
λt=d+a13λt−13+b1Yt−1
관측치들간의 상관성을 고려하기 위해 일반화 선형 모형에서 이전 관측치 Yt−1에 대해 고려하였고 Figure 1을 참조하면 일년마다의 비슷한 패턴이 어느 정도 보여서 조건부 기댓값에 대해서는 t 시점으로부터 13개월 이전 시점 λt−13에 대해 고려하였습니다. R package tscount에서 제공하는 함수 tsglm을 사용해 포아송 조건부 분포와 음이항 조건부 분포로 적합한 결과는 다음과 같습니다.
install.pacakges("tscount")
library(tscount)
## campy
## > Time Series:
## > Start = c(1990, 1)
## > End = c(2000, 10)
## > Frequency = 13
## [1] 2 3 4 1 6 9 12 ... [139] 16 9
########## Model Fitting ##########
campyfit_pois <- tsglm(campy, model=list(past_obs=1, past_mean=13), distr="poisson")
campyfit_nbin <- tsglm(campy, model=list(past_obs=1, past_mean=13), distr="nbinom")
########## Result of Poisson Linear Model ##########
summary(campyfit_pois)[요약통계량]
> Call:
> tsglm(ts = campy, model = list(past_obs = 1, past_mean = 13),
xreg = NULL, distr = "poisson")
> Coefficients:
Estimate Std.Error CI(lower) CI(upper)
(Intercept) 2.296 0.6685 0.9855 3.606
beta_1 0.572 0.0531 0.4680 0.676
alpha_13 0.217 0.0775 0.0648 0.369
Standard errors and confidence intervals (level = 95 %) obtained
by normal approximation.
> Link function: identity
> Distribution family: poisson
> Number of coefficients: 3
> Log-likelihood: -435.4042
> AIC: 876.8084
> BIC: 885.6334
> QIC: 878.0422| "Poisson" | Estimate | Std.Error | CI(lower) | CI(upper) |
| (Intercept) | 2.296 | 0.6685 | 0.9855 | 3.606 |
| beta_1 | 0.572 | 0.0531 | 0.4680 | 0.676 |
| alpha_13 | 0.217 | 0.0775 | 0.0648 | 0.369 |
########## Result of NegBin Linear Model ##########
summary(campyfit_nbin)[요약통계량]
> Call:
> tsglm(ts = campy, model = list(past_obs = 1, past_mean = 13),
xreg = NULL, distr = "nbinom")
> Coefficients:
> Estimate Std.Error CI(lower) CI(upper)
> (Intercept) 2.296 1.0694 0.1998 4.392
> beta_1 0.572 0.0922 0.3916 0.753
> alpha_13 0.217 0.1265 -0.0312 0.465
> sigmasq 0.112 NA NA NA
> Standard errors and confidence intervals (level = 95 %) obtained
by normal approximation.
> Link function: identity
> Distribution family: nbinom (with overdispersion coefficient 'sigmasq')
> Number of coefficients: 4
> Log-likelihood: -406.0332
> AIC: 820.0665
> BIC: 831.833
> QIC: 890.002| "Negative Binomial" | Estimate | Std.Error | CI(lower) | CI(upper) |
| (Intercept) | 2.296 | 1.0694 | 0.1998 | 4.392 |
| beta_1 | 0.572 | 0.0922 | 0.3916 | 0.753 |
| alpha_13 | 0.217 | 0.1265 | -0.0312 | 0.465 |
| sigmasq | 0.112 | NA | NA | NA |
적합 결과를 확인해 보면, 추정된 회귀 계수 값은 같지만 분산이 Yt의 조건부 분포가 음이항 분포인 경우 더 큰 값을 가지는 것을 확인할 수 있습니다. 또한 추정된 과대산포(sigmasq) 값은 0.112로 상대적으로 작은 값을 가집니다. Figure 2를 통해 두 모델의 Yt의 상관계수(Corr(Yt,Yt+h))가 같음을 확인할 수 있으며 계절성이나 상관성이 보이지 않는 것을 확인할 수 있습니다.
########## visualization ##########
plot(campyfit_pois)
plot(campyfit_nbin)
par(mfrow=c(2,1))
acf(residuals(campyfit_pois), main="ACF of Poisson response residuals")
acf(residuals(campyfit_nbin), main="ACF of NegBin response residuals")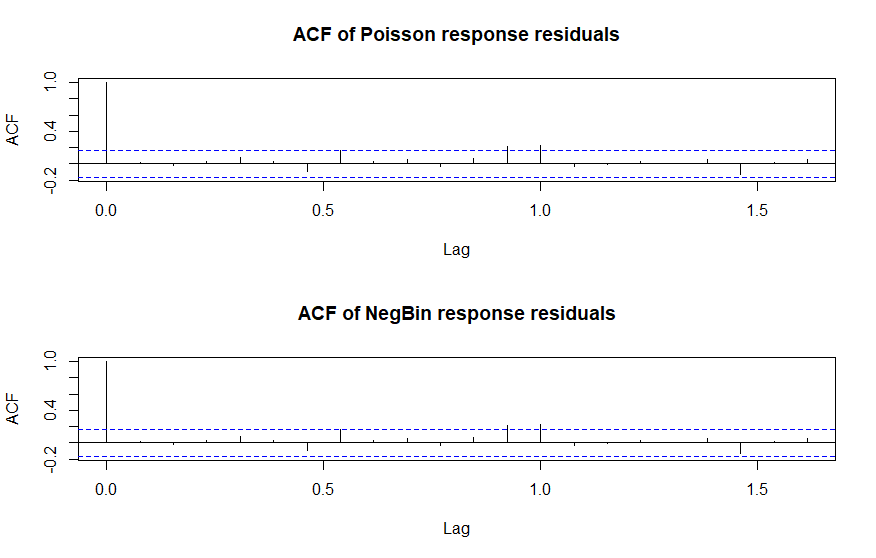
두 개의 모델에 대한 성능 평가 방법은 아래 표와 같으며 결과를 확인해 보면 음이항 분포가 모든 성능 평가 지표에서 더 작은 값을 보이므로 Campy Dataset을 적합할 때는 음이항 조건부 분포를 고려하는 것이 더 타당하다는 사실을 알 수 있습니다.
| Scoring Rule | Abbreviation | Definition |
| logarithmic score | logarithmic | −log(pt(yt) |
| quadratic score | quadratic | −2pt(yt)+||pt||2 |
| spherical score | spherical | −pt(yt)/||pt||2 |
| ranked probability score | rankprob | ∑∞y=0(Pt(y)−1(yt≤y))2 |
| Dawid-Sebastiani score | dawseb | (yt−λt)2/v2t+2log(vt) |
| normalized squared error score | normsq | (yt−λt)2/v2t |
| squared error score & | sqerror | (yt−λt)2) |
rbind(Poisson=scoring(campyfit_pois), NegBin=scoring(campyfit_nbin))
## > logarithmic quadratic spherical rankprob dawseb normsq sqerror
## > Poisson 3.110030 -0.06888556 -0.2618928 2.687020 4.705391 2.3461116 30.92966
## > NegBin 2.900237 -0.07054600 -0.2636117 2.649399 4.136334 0.9785718 30.92966| logarithmic | quadratic | spherical | rankprob | dawseb | normsq | sqerror | |
| Poisson | 3.110030 | -0.06888556 | -0.2618928 | 2.687020 | 4.705391 | 2.3461116 | 30.92966 |
| NegBin | 2.900237 | -0.07054600 | -0.2636117 | 2.649399 | 4.136334 | 0.9785718 | 30.92966 |
Reference
[1].Generalized linear models for count time series: Bosowski, Nicholas and Ingle, Vinay and Manolakis, Dimitris (2017)
[2]. Count time series models: Fokianos, Konstantinos (2012)
[3]. tscount: An R package for analysis of count time series following generalized linear models: Liboschik, Tobias and Fokianos, Konstantinos and Fried, Roland (2017)
'Statistics' 카테고리의 다른 글
| Variational Inference(변분법) (0) | 2024.06.24 |
|---|---|
| Peak Over Threshold(POT)와 Epsilon Threshold (1) | 2024.02.11 |
| Oracle P-values and variable screening (0) | 2022.11.27 |


